Janis Pagel
Erkennung von Protagonisten und Titelfiguren in Dramen
In einem kürzlich erschienenen Artikel haben wir untersucht, inwieweit Protagonisten und Titelfiguren in deutschsprachigen Dramen automatisch erkannt werden können und welche Features einem Machine-Learning-Modell helfen, diese Klassifikation vorzunehmen. Dieser Post kann als Ergänzung zum Artikel gesehen werden.
Inhaltsverzeichnis
- Einrichtung
- Zusatzfunktionen
- Einlesen der Daten
- Trainieren eines Topic Models
- Feature-Extraktion
- Vorbereiten der Daten zur Klassifikation
- Klassifikation
- Evaluation der Ergebnisse
Einrichtung
Um die folgenden Experimente durchzuführen benötigen wir einige Tools und müssen die Daten vorbereiten. Wir stellen unsere Daten zur Verfügung, sodass der Code getestet werden kann. Zunächst benötigen wir die Daten der zu analysierenden Stücke. Wir benutzen die GerDraCor-Daten aus https://github.com/quadrama/data_gdc. Das sind Daten in Tabellenform von unserem GerDraCor-Fork, die mithilfe der DramaNLP-Pipeline erstellt wurden. Falls eigene Daten verwendet werden sollen, sollten sie dasselbe Format wie in https://github.com/quadrama/data_gdc/tree/master/csv. aufweisen.
Wir laden die Daten nun nach R, die Programmiersprache, die wir im Folgenden verwenden werden.
Grundlegende Kenntnis von R ist hilfreich, um den Ausführungen des Posts besser folgen zu können.
Wir benutzen das DramaAnalysis-Paket, um die Daten einzulesen.
Das Paket kann wie auf der Website beschrieben installiert werden. Momentan geschieht dies durch das Ausführen
der folgenden Befehle:
# this is only necessary once per system
install.packages("devtools")
library(devtools)
# Install newest stable version
install_github("quadrama/DramaAnalysis", build_vignettes = TRUE)
Danach können wir die GerDraCor-Daten aus dem QuaDramA-Repository wie folgt installieren:
library(DramaAnalysis)
installData(dataSource = "gdc")
installCollectionData()
Dies installiert die Daten standardmäßig nach QuaDramA/Data2.
Falls dies nicht gewünscht ist, kann der Installationspfad mit dem
setup()-Befehl angepasst werden. Alternativ kann auch die dataDirectory-Option
in installData()1 verwendet werden. Zusätzlich werden wir einen Ordner
mit der Bezeichung data/ verwenden, um dort die trainierten Modelle und andere
generierte Daten zu speichern. Der Ordner kann an beliebiger Stelle erstellt werden
($YOUR_PATH/data). Anschließend kann innerhalb der R-Konsole das aktuelle Verzeichnis
zu diesem Ordner geändert werden mittels setwd($YOUR_PATH).
Wir müssen zusätzliche R-Pakete installieren, um den folgenden Code ausführen zu können:
# Used libraries
library(igraph) # For calculating centrality measures
library(data.table) # Powerful library extending the
# functionality of the build-in
# data.frame type
library(tibble) # Yet another data.frame implementation
library(slam) # Sparse lightweight arrays and matrices
library(topicmodels) # For training custom topic models
library(caret) # Machine learning power house
library(ggplot2) # Advanced plotting
library(iml) # Interpretable machine learning
library(tools) # For title case
Um diese Pakete alle auf einmal zu installieren, kann folgender Befehl verwendet werden:
install.packages(c("igraph", "data.table", "tibble", "slam",
"topicmodels", "caret", "ggplot2", "iml",
"tools"))
Die Pakete müssen anschließend in R geladen werden.
Zusatzfunktionen
Um einige Prozesse besser automatisieren zu können, definieren wir verschiedene hilfreiche Funktione, die weiter unten zur Anwendung kommen.
# Remove the corpus prefix from text IDs
removeCorpusPrefix <- function(str, prefix = "tg") {
sapply(strsplit(str, paste0(prefix,":"), fixed = T),
function(x) (if (is.na(x[2])) {x} else {x[2]}))
}
# The iml package does not make it easy to round the feature
# values for plotting. This function takes the strings of feature
# values coming from iml, transforms them into numbers, rounds them
# and returns the strings with the rounded values.
roundFeatures <- function(featureVector, n_digits = 2) {
features <- lapply(strsplit(featureVector, "="),
function(x) x[1])
values <- round(as.numeric(lapply(strsplit(featureVector, "="),
function(x) x[2])),
digits = n_digits)
paste0(features, "=", values)
}
Dieser Code kann einfach in der R-Konsole ausgeführt werden, um die Funktionen verfügbar zu machen.
Einlesen der Daten
Wir benutzen DramaAnalysis-Funktionen, um die Daten einzulesen.
Da wir die Dramen später auch anhand der Epochen- bzw. Gattungszugehörigkeit
klassifizieren wollen, verwenden wir vordefinierte Kategorien und laden
sie mithilfe von loadSet() (die Listen der Kategorien wurden bereits zuvor mit
installCollectionData() installiert). Danach können wir die eigentlichen Texte
für die kategorisierten Dramen laden2. Da das Laden der kompletten Texte
einige Zeit in Anspruch nimmt, speichern wir die Daten anschließend lokal.
Auf diese Weise können sie in einer späteren Sitzung schnell erneut geladen werden.
# Load text IDs based on their genre
ids.corpora <- list(sd=as.character(loadSet("sturm_und_drang")),
bt=as.character(loadSet(
"buergerliches_trauerspiel")),
wk=as.character(loadSet("weimarer_klassik")),
pop=as.character(loadSet("populaere_stuecke")),
nat=as.character(loadSet("naturalismus")),
wm=as.character(loadSet("wiener_moderne")),
rom=as.character(loadSet("romantik")),
auf=as.character(loadSet("aufklaerung")),
vm=as.character(loadSet("vormaerz"))
)
ids.corpora <- lapply(ids.corpora,
function(x) (gsub("tg", "gdc", x, fixed = TRUE)))
ids.all <- unique(Reduce(c, ids.corpora))
ids.all <- removeCorpusPrefix(ids.all, prefix = "gdc")
# Load the texts
texts <- loadSegmentedText(ids.all, defaultCollection = "gdc")
# Remove rows with unknown speakers
texts <- texts[!is.na(texts$Speaker.figure_surface)]
# Save texts for later use
save(texts, file="data/texts.RData")
Im oben genannten Artikel haben wir sowohl die Erkennung von Protagonisten
als auch von Titelfiguren untersucht. Von einem technischen Standpunkt aus
folgt die Klassifikation von Protagonisten oder Titelfiguren jedoch denselben
Schritten. Im weiteren werden wir uns auf die Annotationen konzentrieren, die
Figuren nach Titlefiguren klassifizieren. Wir lesen die Annotationen von einer
Datei namens titlefigures.csv
ein, die hier heruntergeladen werden kann.
Anschließend die Datei unter $YOUR_PATH/data/titlefigures.csv abspeichern.
Die Datei weist folgende Struktur auf:
Drama1,Titelfigur1,
Drama2,Titelfigur2,Titelfigur3
Drama3,Titelfigur,
...
...
...
d.h. die erste Spalte enthält die Dramen-ID und alle weiteren Spalten enthalten die ID der Titelfigur(en), abhängig von der maximalen Anzahl an Titelfiguren in einem Stück. In unseren Daten wurden nie mehr als zwei Figuren als Titelfiguren genannt, aber abhängig von den verwendeten Daten können auch mehr Spalten verwendet werden. Es ist nur wichtig sicherzustellen, dass die Anzahl an Kommata mit der Maximalanzahl an Spalten übereinstimmt. Die Annotationen werden dann so eingelesen:
heroes <- data.table(read.csv("data/titlefigures.csv",
header = FALSE))
heroes <- melt(heroes, id.vars = "V1", na.rm = TRUE)
heroes$variable <- NULL
heroes <- heroes[value!="",]
colnames(heroes) <- c("drama", "figure")
Zusätzlich zum Einlesen bringen wir die Daten in ein Format, das jede Figur auf einer separaten Zeile listet, zusammen mit der jeweiligen Dramen-ID. Anschließend werden die Spalten zur besseren Lesbarkeit umbenannt.
Trainieren eines Topic Models
Wir haben in unseren Experimenten festgestellt, dass es bessere Ergebnisse liefert, Topic Models auf den Dramendaten zu trainieren, anstatt vortrainierte Daten aus Prosatexten zu verwenden. Wir erreichen dies über folgende Befehle:
ldaModels <- list()
ft <- as.simple_triplet_matrix(as.matrix(frequencytable(texts,
normalize = FALSE)))
ldaModels$lda_5 <- LDA(ft, k = 5, method = "Gibbs")
ldaModels$lda_10 <- LDA(ft, k = 10, method = "Gibbs")
save(ldaModels, file = "data/ldaModels.RData")
Die texts-Variabel enthält die Texte, auf denen wir das Topic Model
trainieren wollen und die bereits in einem vorherigen Abschnitt eingelesen
wurden. Wir benutzen die frequencytable()-Funktionen des DramaAnalysis-Pakets,
um eine Frequenztabelle zu erstellen, die Wortfrequenzen je Drama enthält. Wir speichern
die Tabelle in ft. Danach trainieren wir die Topic Models mithilfe des topicmodels-Pakets.
Hier sind verschiedene Optionen möglich: wir haben uns für Latent Dirichlet Allocation in
Verbindung mit Gibbs sampling entschieden und trainieren zwei Modelle, eines mit fünf
und eines mit zehn Clustern. Anschließend speichern wir die Modelle auf der Festplatte.
Feature-Extraktion
Es ist an der Zeit die Features zu extrahieren, die wir später zum Trainieren des Machine-Learning-Modells verwenden wollen. Dazu laden wir die zuvor eingelesenen Texte und Topic Models und extrahieren alle IDs von den Dramen, für die wir Annotationen haben. Dadurch werden nicht unnötigerweise Features für Dramen extrahiert, die nicht relevant sind.
# Load the texts in case they are not in memory anymore
load("data/texts.RData")
# Load the topic models
load("data/ldaModels.RData")
ids <- ids.all[ids.all %in% unique(heroes$drama)]
Anschließend iterieren wir über alle Texte und speichern alle Figuren
in ein großes data.table-Objekt mit dem Namen p. Dieses data.table-Objekt
enthält eine Liste aller Figuren pro Zeile und listet in den übrigen
Spalten die Figuren-ID sowie alle Features die verwendet werden sollen.
In unserem Fall sind das:
- Anzahl der Figuren pro Stück
- Degree Centrality
- Weighted Degree Centrality
- Betweenness Centrality
- Closeness Centrality
- Eigenvector Centrality
- Wahrscheinlichkeiten einem Topic Model Cluster anzugehören
- Auftritt im letzten Akt
- Anzahl an aktiven Szenen
- Anzahl an passiven Szenen
- Anzahl der geäußerten Tokens
- Zugehörigkeit des Dramas zu einer bestimmten Epoche/Gattung
Die Tabelle enthält auch andere Informationen wie zum Beispiel die Anzahl
an Redeäußerungen pro Figur oder die Token-ID für die erste und letzte
Redeäußerung einer Figur. Die Informationen werden automatisch durch
Funtinonen des DramaAnalysis-Pakets hinzugefügt und im Folgenden nicht
verwendet. Diese Informationen können aber durchaus für zukünftige
Experimente nützlich sein und natürlich können auch weitere neue
Features hinzugefügt werden. Der konkrete Code sieht wie folgt aus:
p <- Reduce(rbind, lapply(ids, function(x) {
# Get a single text
text <- texts[drama==x]
# Create a table with the characters
char <- unique(texts[which(texts$drama==x),
c("corpus",
"drama",
"Speaker.figure_surface",
"Speaker.figure_id")])
# Get the overall number of characters
nfig <- nrow(char)
# Create a presence matrix
pres <- presence(text, passiveOnlyWhenNotActive = TRUE)
# Get the number of tokens a character utters
fstat <- figureStatistics(text)
ft <- frequencytable(text, names = FALSE,
column = "Token.lemma", normalize = FALSE,
byFigure = TRUE)
# Create a co-presence matrix
conf <- configuration(text,by="Scene",onlyPresence = TRUE,
useCharacterId = TRUE)
copr <- conf$matrix %*% t(conf$matrix)
rownames(copr) <- conf$figure
colnames(copr) <- conf$figure
# Find out if a character appeared in the last act
confAct <- configuration(text, by="Act", onlyPresence = TRUE,
useCharacterId = TRUE)
lastAct <- cbind(confAct$drama, figure = confAct$figure,
lastAct = as.numeric(confAct$matrix[,
ncol(confAct$matrix)]))
# Get a co-presence graph based on the co-presence matrix
g <- graph_from_adjacency_matrix(copr, mode = "upper",
diag = FALSE, weighted = TRUE)
# Calculate different centrality measures
dg <- degree(g, normalized = TRUE)
wdg <- strength(g)
bt <- betweenness(g, normalized = TRUE)
cn <- closeness(g, normalized = TRUE)
ev <- eigen_centrality(g)
# Get the posterior probability for a character to be part
# of a certain topic, based on their uttered tokens
post <- posterior(ldaModels$lda_10, ft)$topics
post.df <- data.frame(figure = substr(rownames(post),
8,
100),
post)
# Merge all features into a feature table with each character
# as a row
pres <- merge(pres, char,
by.x = c("corpus", "drama", "figure"),
by.y = c("corpus", "drama", "Speaker.figure_id"))
pres$nfig <- rep(nfig, nrow(pres))
pres <- merge(pres, lastAct)
pres <- merge(pres, fstat)
pres <- merge(pres, post.df, by = c("figure"))
pres <- merge(pres, data.frame(degree = dg, figure = names(dg)))
pres <- merge(pres, data.frame(wdegree = wdg,
figure = names(wdg)))
pres <- merge(pres, data.frame(between = bt, figure = names(bt)))
pres <- merge(pres, data.frame(close = cn, figure = names(cn)))
pres <- merge(pres, data.frame(eigen = ev$vector,
figure = names(ev$vector)))
# Return the feature table
pres
}))
Nachdem p erstellt wurde, nehmen wir noch einige Änderungen vor. Die
drama-Spalte sollte zu Faktoren transformiert werden. Um eine bessere
Lesbarkeit zu erreichen, benennen wir zudem die Spalten um, die durch das
Topic Model automatisch erstellt wurden, d.h. X wird zu T (für Topic).
Die Informationen darüber, welches Drama zu welcher Epoche/Gattung gehört, ist
nicht von der einzelnen Figur abhängig und kann jetzt eingefügt werden.
Zum Schluss müssen wir noch herausfinden, ob eine Figur nach unseren
Annotationen Titelfigur ist oder nicht. Dazu benutzen wir die
zuvor erstellte heroes-Tabelle und fügen die Information in einer Spalte
class hinzu. Falls die Figur tatsächlich Titelfigur ist, erhält sie die
Markierung TF (Titelfigur), andernfalls NTF (Keine Titelfigur). Optional
können wir auch die Featuretabelle zur späteren Verwendung speichern.
p$drama <- factor(p$drama)
names(p) <- gsub("X", "T", names(p), fixed = TRUE)
p$SD <- as.integer(paste(p$corpus, p$drama, sep = ":")
%in% ids.corpora$sd)
p$BT <- as.integer(paste(p$corpus, p$drama, sep = ":")
%in% ids.corpora$bt)
p$WK <- as.integer(paste(p$corpus, p$drama, sep = ":")
%in% ids.corpora$wk)
p$POP <- as.integer(paste(p$corpus, p$drama, sep = ":")
%in% ids.corpora$pop)
p$NAT <- as.integer(paste(p$corpus, p$drama, sep = ":")
%in% ids.corpora$nat)
p$WM <- as.integer(paste(p$corpus, p$drama, sep = ":")
%in% ids.corpora$wm)
p$ROM <- as.integer(paste(p$corpus, p$drama, sep = ":")
%in% ids.corpora$rom)
p$AUF <- as.integer(paste(p$corpus, p$drama, sep = ":")
%in% ids.corpora$auf)
p$VM <- as.integer(paste(p$corpus, p$drama, sep = ":")
%in% ids.corpora$vm)
p$class <- ifelse(apply(p, 1, function(x) (if (any(x["drama"] ==
heroes$drama & x["figure"] == heroes$figure))
{TRUE} else {FALSE})),
"TF",
"NTF")
p$class <- factor(p$class)
save(p, file = "data/p.RData")
> head(p)
figure corpus drama scenes actives passives
1: andreas gdc tzgk.0 75 5 13
2: arabella gdc tzgk.0 75 5 2
3: asserato gdc tzgk.0 75 3 2
4: bedienter gdc tzgk.0 75 1 2
5: bella gdc tzgk.0 75 2 8
6: berta gdc tzgk.0 75 4 6
presence Speaker.figure_surface nfig lastAct
1: -0.10666667 ANDREAS 38 1
2: 0.04000000 ARABELLA 38 1
3: 0.01333333 ASSERATO 38 0
4: -0.01333333 BEDIENTER 38 0
5: -0.08000000 BELLA 38 0
6: -0.02666667 BERTA 38 1
length tokens types utterances utteranceLengthMean
1: 32469 820 406 25 32.800000
2: 32469 288 160 21 13.714286
3: 32469 149 100 15 9.933333
4: 32469 10 10 1 10.000000
5: 32469 104 81 3 34.666667
6: 32469 212 111 32 6.625000
utteranceLengthSd firstBegin lastEnd T1
1: 25.734218 77418 196198 0.03171953
2: 12.810710 3603 189928 0.06400000
3: 4.511361 62813 144980 0.04697987
4: NA 152575 152646 0.12500000
5: 32.129944 75759 154735 0.07500000
6: 3.687380 35983 197145 0.16756757
T2 T3 T4 T5
1: 0.04006678 0.02838063 0.03672788 0.2520868
2: 0.03200000 0.03600000 0.06800000 0.0760000
3: 0.04026846 0.04026846 0.05369128 0.2080537
4: 0.08928571 0.08928571 0.08928571 0.1071429
5: 0.05833333 0.05833333 0.06666667 0.3250000
6: 0.05405405 0.04864865 0.05405405 0.1621622
T6 T7 T8 T9
1: 0.01001669 0.5292154 0.02337229 0.02671119
2: 0.02400000 0.5680000 0.03600000 0.04400000
3: 0.03355705 0.4362416 0.04697987 0.04697987
4: 0.08928571 0.1428571 0.08928571 0.08928571
5: 0.05000000 0.2083333 0.05833333 0.05000000
6: 0.05945946 0.3027027 0.06486486 0.04864865
T10 degree wdegree between
1: 0.02170284 0.16216216 8 0.0193078793
2: 0.05200000 0.21621622 11 0.1005694872
3: 0.04697987 0.24324324 15 0.0516348166
4: 0.08928571 0.02702703 1 0.0000000000
5: 0.05000000 0.05405405 4 0.0000000000
6: 0.03783784 0.10810811 7 0.0005744381
close eigen SD BT WK POP NAT WM ROM AUF
1: 0.4512195 0.069323313 1 0 0 0 0 0 0 0
2: 0.4868421 0.105234584 1 0 0 0 0 0 0 0
3: 0.4252874 0.251059554 1 0 0 0 0 0 0 0
4: 0.3557692 0.026868517 1 0 0 0 0 0 0 0
5: 0.2242424 0.008853292 1 0 0 0 0 0 0 0
6: 0.3814433 0.164283361 1 0 0 0 0 0 0 0
VM class
1: 0 NTF
2: 0 NTF
3: 0 NTF
4: 0 NTF
5: 0 NTF
6: 0 NTF
Vorbereiten der Daten zur Klassifikation
We nehmen nun einige letzte Anpassungen vor, bevor wir das Model
mit den extrahierten Features trainieren. Wir normalisieren die Features
actives, passives und tokens anhand der Gesamtlänge eines Dramas.
Um besser lesbare Plots zu erhalten, transformieren wir zudem die Figurenbezeichnungen
in ein schöneres Format. Danach erstellen wir ein Backup der originalen Tabelle
und entfernen alle Informationen, die für das Machine Learning nicht
relevant sind. Wir speichern die originale und die Trainingstabelle in die
Variabel pTF.
prepareData <- function(p) {
p[is.na(p)] <- 0
p <- na.omit(p)
p$actives <- p$actives/p$scenes
p$passives <- p$passives/p$scenes
p$tokens <- p$tokens/p$length
p$presence <- NULL
p$Speaker.figure_surface <- toTitleCase(tolower(
as.character(
p$Speaker.figure_surface)))
orig <- p
p$corpus <- NULL
p$figure <- NULL
p$length <- NULL
p$scenes <- NULL
p$Speaker.figure_surface <- NULL
p$drama <- as.integer(p$drama)
list(p = p, orig = orig)
}
pTF <- prepareData(p)
Klassifikation
Zunächst definieren wir die Machine-Learning-Methode, die verwendet werden soll.
Wir benutzen hier einen Random-Forest-Classifier aus dem caret-Paket.
Die Funktion testFeatureSet() ermöglicht es, jedesmal denselben Seed zu verwenden,
wenn die caret-Methode aufgerufen wird. Wir können auch die Sampling-Methode und die
Features als Argumente in die Funktion geben. Die Ausgabe ist eine Liste, die
das trainierte Modell enthält, sowie die Eingabe und Ausgabe des Modells, eine Konfusionsmatrix der Ergebnisse,
die Feature-Importance-Werte, die konkreten Vorhersagen für einzelne Figuren sowie
richtig und falsch vorhergesagte Figuren. Wie können auch bereits die Kombination aus Features
definieren, die wir später testen wollen. Die Bezeichnungen der Features sind einfach
die Spaltennamen aus pTF.
# Define which classifier to use
caretMethod <- "rf"
# Define the number of sampling repeats
repeatsVariable <- 10
# Set seed for the random number generator
seed <- 42
set.seed(seed)
testFeatureSet <- function(features, p, orig, sampling, seed = 42) {
set.seed(seed)
fv <- as.matrix(subset(p, select = -c(class)))
x <- fv[, features, drop = FALSE]
x <- as.data.frame(x)
control <- trainControl(method = "repeatedcv",
number = 10,
repeats = repeatsVariable,
savePredictions = "all",
sampling = sampling,
classProbs = TRUE)
model <- train(x = x,
y = p$class,
method = caretMethod,
trControl = control,
preProc = c("center", "scale"))
if (length(features) > 1) {
varI <- varImp(model, scale = FALSE)
} else {
varI <- data.frame()
}
predictions <- predict(model, x)
cm <- list(TF = confusionMatrix(predictions,
p$class,
mode = "everything",
positive = "TF"),
NTF = confusionMatrix(predictions,
p$class,
mode = "everything",
positive = "NTF"))
pred <- data.frame(orig[,c("corpus", "drama", "figure", "BT",
"WK", "SD", "POP", "NAT",
"WM", "ROM", "AUF", "VM")],
pred = predictions, ref = orig[, "class"])
falsePredictions <- pred[pred$pred != pred$class, ]
correctPredictions <- pred[pred$pred == pred$class, ]
list(model = model, x = x, y = p$class, imp = varI, cm = cm,
pred = pred, correct = correctPredictions,
mistakes = falsePredictions)
}
features <- c(c("BT" ,"WK", "SD", "POP", "NAT", "WM", "ROM",
"AUF", "VM"),
"tokens",
c("degree", "wdegree", "close", "eigen", "between"),
c("actives", "passives"),
paste0("T",1:10),
"nfig",
"lastAct")
featuresWithoutTokens <- c(c("BT" ,"WK", "SD", "POP", "NAT",
"WM", "ROM", "AUF", "VM"),
c("degree", "wdegree", "close",
"eigen", "between"),
c("actives", "passives"),
paste0("T",1:10),
"nfig",
"lastAct")
Jetzt rufen wir einfach testFeatureSet() mit verschiedenen Feature-Kombinationen
und der gewünschten Sampling-Methode auf. Wir können zum Beispiel als eine einfache
Baseline nur das tokens-Feature benutzen.
Wir trainieren auch zwei weitere Modelle: eines mit allen Features außer dem Tokens-Feature,
sowie ein Modell mit allen Features. Wir verwenden die Sampling-Methode smote und
speichern die Modelle nach dem Training ab.
results <- list()
results$TokensBL <- testFeatureSet("tokens",
pTF$p,
pTF$orig,
sampling = "smote",
seed)
results$woTokens <- testFeatureSet(featuresWithoutTokens,
pTF$p,
pTF$orig,
sampling = "smote",
seed)
results$All <- testFeatureSet(features,
pTF$p,
pTF$orig,
sampling = "smote",
seed)
save(results, file = "data/results.RData")
Evaluation der Ergebnisse
Wir können die Modelle auf verschiedene Weisen evaluieren3:
- Konfusionsmatrix der vorhergesagten Klassen gegenüber den tatsächlichen Klassen
- Feature Importance
- Vorhersagen für einzelne Figuren
Beginnen wir mit der Konfusionsmatrix. Wir können die Konfusionsmatrix für ein
spezifisches Modell direkt vom Output von testFeatureSet() beziehen:
cm <- results$All$cm$TF$table
> cm
Reference
Prediction NTF TF
NTF 1100 0
TF 66 42
Als nächstes werden wir auch verschiedene Evaluationsmetriken auf Basis der Konfusionsmatrizen berechnen, wie etwa Precision, Recall, F1 und Accuracy. Dies kann für alle Modelle auf einmal angewandt werden, um einen direkten Vergleich zu ermöglichen:
dfTF <- data.frame(lapply(results, function(x) {x$cm$TF$byClass}))
dfNTF <- data.frame(lapply(results, function(x) {x$cm$NTF$byClass}))
dfO <- data.frame(lapply(results, function(x) {x$cm$NTF$overall}))
dfEval <- rbind(dfTF, dfNTF, dfO)
dfEval <- dfEval[c("Precision", "Recall", "F1", "Accuracy"),]
> dfEval
TokensBL woTokens All
Precision 0.3387097 0.3281250 0.3888889
Recall 1.0000000 1.0000000 1.0000000
F1 0.5060241 0.4941176 0.5600000
Accuracy 0.9321192 0.9288079 0.9453642
Die Feature Importance können wir auch direkt auslesen und in folgender Weise plotten:
imp <- results$All$imp$importance %>%
as.data.frame() %>%
rownames_to_column() %>%
ggplot(aes(x = reorder(rowname, Overall), y = Overall)) +
geom_bar(stat = "identity", alpha = 0.8) +
coord_flip() +
labs(x = "Feature", y = "Importance")
> imp
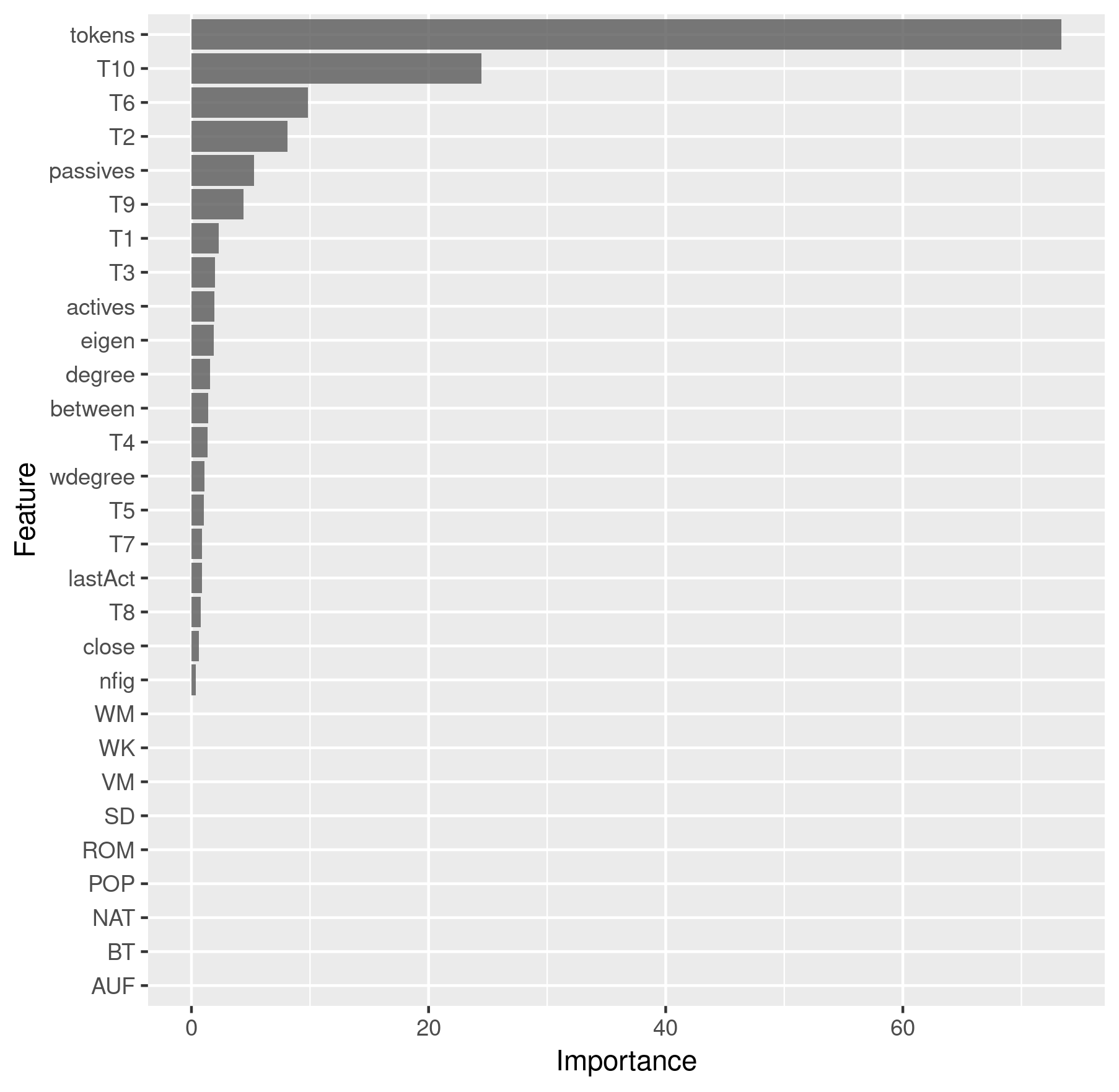
Feature Importance
Schlussendlich können wir die Vorhersagen eines Modells für eine spezifische Figur untersuchen. Dies kann besonders interessant sein, wenn wir vermuten, dass eine Figur besonders schwierig zu klassifizieren ist und wir können auch die Vorhersagen für mehrere Figuren, z.B. aus dem selben Stück, anzeigen und vergleichen. Wir nehmen Emilia und Marinelli aus Lessings Emilia Galotti als Beispiel. Natürlich sind wir auch daran interessiert, ob das Modell die richtige Klassifizierung für diese beiden Figuren vorgenommen hat:
chars <- c("emilia", "marinelli")
predChar <- results$All$pred[results$All$pred$figure %in% chars &
results$All$pred$drama == "rksp.0", ][,
c("corpus", "drama", "figure", "class", "pred")]
> predChar
corpus drama figure class pred
gdc rksp.0 emilia TF TF
gdc rksp.0 marinelli NTF TF
Wie wir sehen, wurde Emilia richtig als Titelfigur erkannt, während Marinelli fälschlicherweise auch als Titelfigur klassifiziert wurde, was aber nicht zutrifft.
Zum Schluss führen wir eine Shapley-Analyse durch, die die wichtigsten
Features für einen einzelnen Datenpunkt wiedergibt. Je höher der phi-Wert
ist, desto wichtiger war das Feature für die Klassifikation. Negative
phi-Werte deuten darauf hin, dass das jeweilige Feature sogar kontraproduktiv
für die Klassifikation der jeweiligen Klasse war.
Wir benutzen das iml-Paket
für die Shapley-Analyse. Zunächst erstellen wir einen iml-Prädiktor:
x <- results$All$x
y <- results$All$y
predictor <- Predictor$new(results$All$model, data = x, y = y)
Dann können wir darauf basierend die Shapley-Werte bestimmen und plotten:
charID <- rownames(results$All$pred[
results$All$pred$figure == "emilia"
& results$All$pred$drama == "rksp.0", ])
shapley <- Shapley$new(predictor, x.interest = x[charID, ])
# Round the feature values so that they don't clutter the plot
shapley$results$feature.value <-
roundFeatures(shapley$results$feature.value)
shapley_emilia <- shapley$results[
which(shapley$results$class == "TF"), ] %>%
ggplot(aes(x = reorder(feature.value, -phi), y = phi)) +
geom_bar(stat = "identity", alpha = 0.8) +
coord_flip() +
labs(x = "Feature", y = "phi")
charID <- rownames(results$All$pred[
results$All$pred$figure == "marinelli"
& results$All$pred$drama == "rksp.0", ])
shapley$explain(x.interest = x[charID, ])
shapley$results$feature.value <-
roundFeatures(shapley$results$feature.value)
shapley_marinelli <- shapley$results[
which(shapley$results$class == "TF"), ] %>%
ggplot(aes(x = reorder(feature.value, -phi), y = phi)) +
geom_bar(stat = "identity", alpha = 0.8) +
coord_flip() +
labs(x = "Feature", y = "phi")
> shapley_emilia
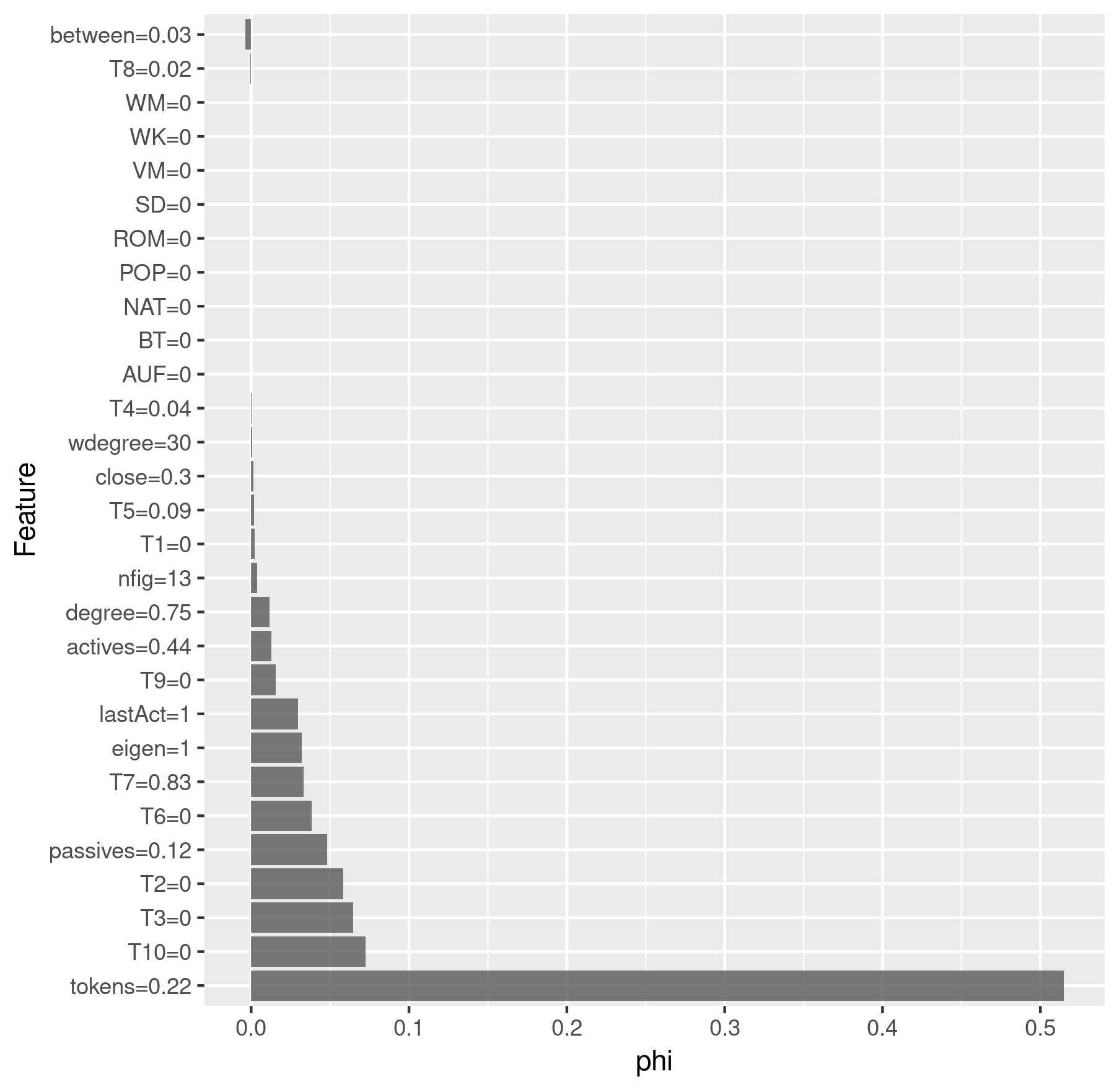
Shapley-Analyse für Emilia
> shapley_marinelli
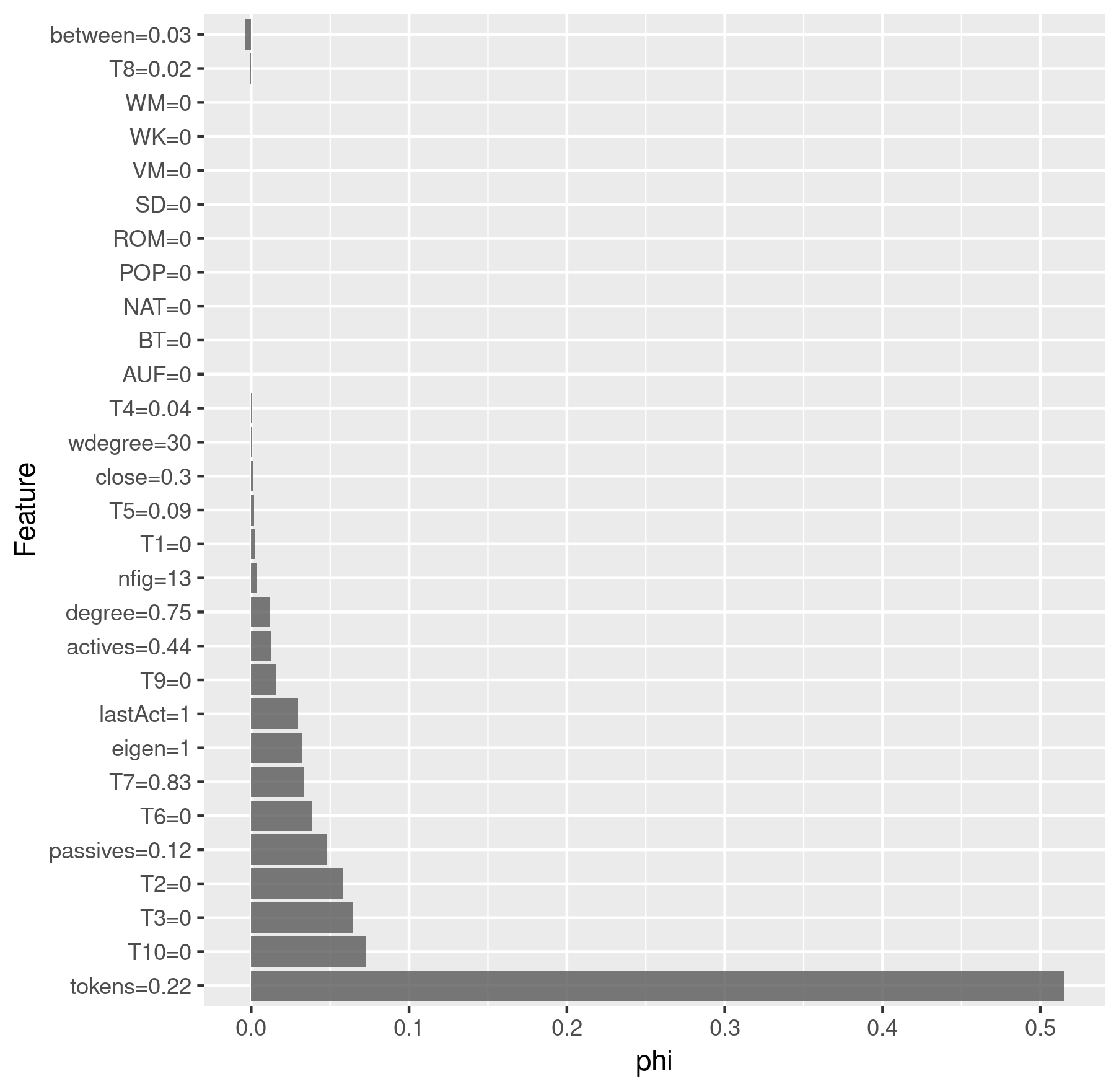
Shapley-Analyse für Marinelli
Das ist alles. Der komplette Code ist hier verfügbar.
-
Für eine ausführliche und aktuelle Beschreibung des Installationsprozesses, siehe das
DramaAnalysisWiki. ↩ -
Da die Kategorisierungen momentan nur für TextGrid verfügbar sind, werden einige nicht geladen, falls sie nicht in GerDraCor verfügbar sind. Die Fehlermeldungen, die R dafür ausgibt, können ignoriert werden. ↩
-
Die Implementation von Punkt 2 und 3 wurde inspiriert durch https://www.r-bloggers.com/interpretable-machine-learning-with-iml-and-mlr/ und https://shirinsplayground.netlify.com/2018/07/explaining_ml_models_code_caret_iml/. Auf diesen Seiten finden sich noch mehr Anregungen zur Interpretation von Blackbox-Machine-Learning-Modellen in R. ↩